目次
はじめに
はじめまして、神戸大学大学院人間発達環境学研究科の徐寿明です。
突然ですが、皆さんは「環境DNA」という言葉を聞いたことはありますか?
僕は大学生になって初めてこの言葉を知り、魅せられ、気づけばもう8年が経ちます。詳しいことは後述しますが、この環境DNAは、今までの生物モニタリング手法の常識を大きく覆しました。この記事では、そんな環境DNAのことについて、僕自身の研究も踏まえながら紹介していきます。
研究背景
生物多様性を守るために
昨今の地球環境問題の中で、特に深刻視されているのが「生物多様性の喪失」です。人間活動の影響などにより、地球上から多くの生き物が (私たちの知らない間に) その姿を消しつつあります。
生物多様性とは、それ自体に大きな価値があるだけでなく、生物多様性を始めとした生態系から得られる様々な恵み (生態系サービス) によって私たちの生活は支えられており、生物多様性の保全は私たち人類にとって非常に重要なテーマです。
生物多様性の保全のために、生き物の分布、つまりどこにどんな生き物がいるかを知る必要があります。それも、なるべく広範囲かつ迅速なモニタリングが求められます。例えば、皆さんの家の近くの池や川にどんな生き物がいるのか、どうやって調べるのか考えてみましょう。釣りや手網、罠を仕掛けたり潜ったり (某TVでは池の水を抜いたり、、、)、色々な方法があると思います。
しかし、これら従来の方法は、幾つかの問題を抱えています。
- 生き物やその生息地にダメージを与えたり、場合によっては死なせてしまう
- 例えば釣りが上手な人とそうでない人の間で、結果が変わってしまう
- 見た目での分類の判断にも、専門的な知識が必要 (場合によっては分類ができない)
などなど。こうした問題を全て解決できるとして近年大きく注目されているのが、この記事のテーマである環境DNAを用いたモニタリングなのです。
環境DNA分析とは
一言で表せば、環境DNAとは「生き物の痕跡」です。いわば化石や足跡のようなもので、生き物がいた痕跡を、DNA情報から読み解くのです。
例えば魚や両生類なら、排泄物や粘液などの形で自らのDNAを常に水中に放出していると考えられています (他の生き物でも同様に、皮膚や毛、唾液や汗など)。こうした環境中にDNAとして存在する生き物の痕跡を、環境DNAと呼びます。
この環境DNA分析の大きな特徴は、水を汲み、その中に含まれるDNAを調べることで、生き物の分布を推測できるということです。したがって、
- 生き物や生息地にダメージを全く与えない (非侵襲的)
- 調査者間で結果が大きく変わらない
- 塩基配列は種によって異なるので、形態学的な専門知識を必要としない
などの利点があり、従来の方法よりも簡便かつ高感度なモニタリングが可能なのです。
環境DNA分析による生物モニタリングの試みは、ヨーロッパから始まりました。フランスのため池で、見たり捕まえたりすることなしに、外来種であるウシガエルの在不在や生物量を、水を汲むだけで推測できることを示したのが、初めての報告例でした1。それから十数年が経ち、今や環境DNA分析は全世界で注目を浴びる新技術の一つとなりました。
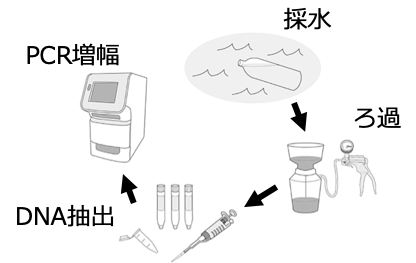
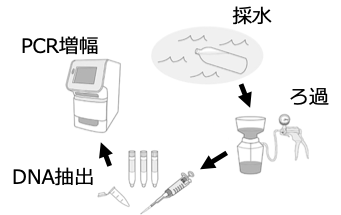
もう少し具体的に、環境DNA分析の手順についてお話しましょう (図1)。水を汲み、フィルターでろ過することにより、水の中に溶けている環境DNAをフィルターに回収します。そして、そのろ過フィルターからDNAを抽出し、対象とする生き物のDNAをPCR法 (ポリメラーゼ連鎖反応; 特定のDNA断片のみを増幅させることができる技術) によって分析します。したがって、水サンプル中のDNAがPCR増幅すれば、対象の生き物はその場所におり、増幅しなければ生き物はそこにいない、というように推測するわけです。
環境DNA分析の適用例
幾つかの研究例を紹介して、環境DNA分析によって具体的にどんなことができるのか、お話しましょう。
事例1. オオサンショウウオの分布モニタリング
オオサンショウウオは、日本に生息する世界最大の両生類で、国の特別天然記念物にも指定されています。しかし近年、外来種であるチュウゴクオオサンショウウオとの交雑により、国内のオオサンショウウオの遺伝子汚染 (≒オオサンショウウオに固有の遺伝子が失われる) が懸念されています。
そこで、そもそもオオサンショウウオとチュウゴクオオサンショウウオがどこに生息しているのかを知る必要があるのですが、彼らは夜行性かつ水の中で暮らすので、見つけるのがとても大変です。そこで、環境DNA分析を用いて、京都府の加茂川・桂川水系におけるオオサンショウウオの分布調査を行ったのでした。
環境DNA分析は、たった8日間で、オオサンショウウオとチュウゴクオオサンショウウオの季節的な分布調査を可能にしただけでなく、チュウゴクオオサンショウウオの侵入状況を明らかにしました2 (図2a)。
この研究の最も重要な点は、これまでの捕獲調査で知られていたよりも、チュウゴクオオサンショウウオの分布が拡大している可能性を示したことです。希少種の保全管理や外来種の早期発見にも、環境DNA分析は大きく貢献することができます。
事例2. 魚類の網羅的な検出
環境DNA分析では、特定の生き物の分布調査だけではなく、複数の生き物の分布をまとめて調べることもできます。これを、環境DNAメタバーコーディング法と呼びます。ある場所における生き物の組成や多様性を知りたい場合、メタバーコーディングは非常に強力なツールとなります。
2014年、日本の研究者たちは、沖縄県の美ら海水族館で飼育している魚の種数を、たった10 Lの水槽水からどれくらい推測できるのかを調べました。その結果、環境DNAメタバーコーディングは、全魚種の93.3 % (168種) を言い当てることに成功したのです3 (水槽によっては100 % の一致率!) (図2b)。
また、水族館近くの海からも水を汲み、たった10 Lの海水から93種もの魚のDNAを検出することができました。環境DNA分析がいかに効率的かつ高感度なモニタリングツールであるか、お分かり頂けたでしょうか?

環境DNA分析に残された課題
さて、ここまで環境DNA分析の長所について触れてきましたが、「生き物の痕跡」であるが故に以下のような課題もあります。
(1) 他の方法と同様に、そこに生き物がいれば必ず100 % 検出できるわけではない
(2) 検出された環境DNAが、いつどこで元の生き物から放出されたのか分からない時がある
例えば、ずいぶん前に放出された環境DNAや、死亡個体に由来する環境DNAを検出してしまうと、その結果に基づく推測は不確かなものになってしまいます。
つまり、環境DNAが生物モニタリングにおける有用なツールであるためには、どれくらいの時空間的な範囲で、環境DNAが生き物の分布を示すのかを知る必要があります。
もしも環境DNAが示す時空間的な生物情報を調節することができ、例えば、生き物から放出されて間もない「新鮮な環境DNA」を選択的に回収することができれば、環境DNAに基づく推測精度の改善につながるでしょう。いわば「環境DNAの時間を操れるか?」、僕の卒業研究はそんなテーマから始まりました。
本論
環境DNAの時間を操るために
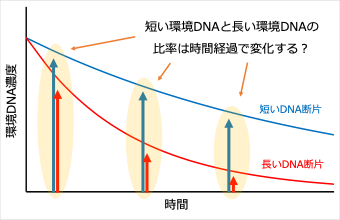
この問いに答えるために、僕が着目したのは「DNA断片長と分解率の関係」でした。環境DNAは生き物から放出された後、時間経過に伴って次第に検出されなくなります。これは、微生物などの働きによってDNAの切断や欠損が起こり、PCRで検出できるDNA断片の量が減っていくからです。
したがって、PCRの際に長いDNA断片をターゲットにした方が、そのサンプルから対象のDNAを検出できる時間は、より短くなると考えられます。つまり、より長いDNA断片ほど、より短時間で検出できなくなる、「長い環境DNAほど早く分解される (=分解率が高い)」と考えたのです (図3)。
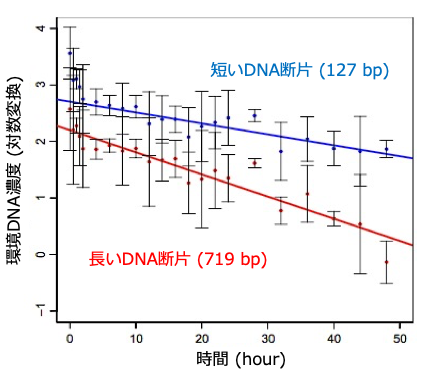
この仮設を検証するために、僕は水槽実験を行いました。2日間にわたってマアジ (Trachurus japonicus) の飼育水を水槽から採水し続け、その中に含まれる短い環境DNAと長い環境DNAの分解率を比較しました。その結果、長いDNA断片の方が、確かに高い分解率を示したのです4 (図4)。
より断片長の大きい環境DNAほど野外での検出量が低くなることは分かっていましたが、これがDNA断片長間での分解率の違いに起因することを、この結果は世界で初めて示しました。
次に僕は、この残存性の違いを用いて、野外でどんなことができるのかに興味を持ちました。京都府北部の舞鶴湾に生息するマアジを対象に、以下のような実験を行いました5。
(1) 湾内47地点を船で回り、表層から海水を採取する (これだけのサンプリングが日中には全て終わります)。
(2) 同時に、魚群探知機を用いて、その地点におけるマアジの生物量を推定する。
(3) 海水サンプルに含まれるマアジ環境DNA濃度を、短いDNA断片と長いDNA断片についてそれぞれ推定し、それぞれの断片長の環境DNAの分布と、魚探に基づくマアジの分布を比較する。
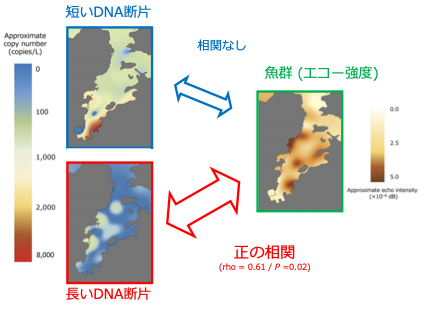
その結果、短いDNA断片に着目すると、環境DNA濃度は実際のマアジの分布とあまり相関しなかったのに対して、長いDNA断片に着目すると、環境DNA濃度はマアジの分布と正の相関を示すことが分かりました4 (図5)。
環境DNAに基づく生物量推定における長いDNA断片の有用性が示されたわけですが、一方で疑問が残ります。何故、短いDNA断片では、環境DNAと生物量の相関が見られなくなったのでしょうか?
長い環境DNAができたこと、短い環境DNAができなかったこと
幾つかの先行研究6は、環境DNA濃度と生物量が正に相関することを示しており、それらのほとんどは、100-200 bp (bp: 塩基長の単位; base pair) 程度の短い環境DNAを対象としています。したがって、長い環境DNAはともかく、短い環境DNAの分布が生物量と相関しないのは不自然でした。
短いDNA断片に基づいて推定されたマアジ環境DNAの湾内分布を見てみると、ほとんどのマアジは右下の赤色の部分に集中して生息していることになります (図5)。しかし、魚群探知機に基づくマアジの湾内分布と比較してみると、実はこれらの分布は少しずれていて、少なくとも短い環境DNAが最も多く検出された地点で、マアジの生息はほとんど確認できないのです。
なぜ、いるはずのないマアジのDNAが、こんなにも多く検出されてしまったのでしょうか?
実は、この地点のすぐ近くには、大きな漁港があり、マアジを含めた色んな魚介類が毎日引揚げられています。つまり、この地点で検出されたマアジの環境DNAは、生きている個体ではなく「死んだ個体」に由来した、時間が経ち分解の進んだ環境DNAであると考えられるわけです。
こうした古い環境DNAは、放出されて間もない環境DNAに比べて、そのDNA断片長が短くなっているであろうことは、ここまでの話から想像に難くないでしょう。実際、この短い環境DNAが最も多く検出された地点で、長い環境DNAは何と全く検出されませんでした。
長い環境DNAにできて、短い環境DNAにできなかったこと、それは「古い環境DNAの影響をキャンセルし、時間的により新しい環境DNAを選択的に検出する」ことだったのです。特に今回の調査地のような環境では、この長い環境DNAの長所が存分に活かされることとなりました。
終わりに
生態系の持続的な利用や環境保全など、「環境DNA学を人類全体の幸福に資する学問分野として育成・発展させる」ことを目的として、2018年には一般社団法人 環境DNA学会 (The eDNA Society) が設立されました。日本のみならず世界各国で、環境DNA分析の社会実装に向けて産・官・学が連携して様々なことを進めています。
そのために今求められているのは、やはり環境DNAの基礎的な知見 (性質、動態、メカニズム) です。環境DNAは何に由来し、どんな状態で、どれくらい環境中で移動し、どれくらい長く検出され続けるのか、など、こうした知見の一つ一つが環境DNA分析の発展につながります。
これからも、こうした環境DNAの基礎情報の理解を深めることを通して、環境DNA学に貢献できればと思います。最後までお読みいただき、誠にありがとうございました。
引用文献
(1) Ficetola, G. F., Miaud, C., Pompanon, F., & Taberlet, P. (2008). Species detection using environmental DNA from water samples. Biology Letters, 4(4), 423-425.
(2) Fukumoto, S., Ushimaru, A., & Minamoto, T. (2015). A basin‐scale application of environmental DNA assessment for rare endemic species and closely related exotic species in rivers: a case study of giant salamanders in Japan. Journal of Applied Ecology, 52(2), 358-365.
(3) Miya, M., Sato, Y., Fukunaga, T., Sado, T., Poulsen, J. Y., Sato, K., … & Kondoh, M. (2015). MiFish, a set of universal PCR primers for metabarcoding environmental DNA from fishes: detection of more than 230 subtropical marine species. Royal Society Open Science, 2(7), 150088.
(4) Jo, T., Murakami, H., Masuda, R., Sakata, M. K., Yamamoto, S., & Minamoto, T. (2017). Rapid degradation of longer DNA fragments enables the improved estimation of distribution and biomass using environmental DNA. Molecular Ecology Resources, 17(6), e25-e33.
(5) Yamamoto, S., Minami, K., Fukaya, K., Takahashi, K., Sawada, H., Murakami, H., … Kondoh, M. (2016). Environmental DNA as a ‘snap‐ shot’ of fish distribution: A case study of Japanese jack mackerel in Maizuru Bay, Sea of Japan. PLoS ONE, 11(3), e0149786.
(6) Takahara, T., Minamoto, T., Yamanaka, H., Doi, H., & Kawabata, Z. (2012). Estimation of fish biomass using environmental DNA. PLoS ONE, 7(4), e35868.