神戸大学大学院 人間発達環境学研究科 人間環境学専攻 修士課程2年の速水花奈と申します。
生態学系の研究室に所属しており、その中でも「環境DNA」という1つのモニタリング手法に着目した研究を行なっています。
皆さんの中に、「環境DNA」という言葉を聞いたことがある人はいますか?
あとで詳しくお話しますが、簡単に言うと、生物が環境中に何らかの形で放出しているDNAのことです。
よく刑事ドラマで、DNA鑑定をして犯人を特定するシーンがありますよね?(最近だとアンナチュラルやトレースなどです)
そんな現場の痕跡からDNA情報を得ることで犯人を特定する鑑識調査のように、水中に漂っているDNAを回収・分析することでそこにすむ生き物を特定する技術が環境DNA分析なのです。
この記事では、そんな環境DNA分析の魅力を私自身の研究と共に紹介していきたいと思います。
目次
はじめに
水の中の生き物を知る方法
さっそくですが、皆さんに質問です。
水の中にどんな生き物がいるか知りたければ、どのような方法を用いて調べますか?
実際に、水の中の生き物を知る方法は色々あると思います。
例えば
釣りをする、潜って目視で観察する(潜水目視)、網を投げて捕まえる(投網, タモ網, 手網など)、他にも電気ショッカーや魚群探知機を使用して調査する
などの方法があげられます。
これまでは、このような手法を用いた調査が一般的でした。
しかし、これらの手法を用いて水中の生き物を把握するには、多大な労力とコストが必要です。さらに、
- 地形や植生によっては網が打てない
- 水の濁りなどで目視できない
- 穴や石の下に潜り込んでいて捕獲できない
といった問題もしばしば生じます。そして何より、その捕まえたもしくは目視で確認した種が何の種であるのかを同定できる専門家が必要不可欠となってきます。
では、どうすればいいのか…
そこで登場したのが『環境DNA分析手法』なのです。
そもそも環境DNAとは何なのか?
環境DNAとはエラや表皮から剥がれ落ちた細胞片や、糞や粘液などに由来する体外に放出されたDNAの総称です。
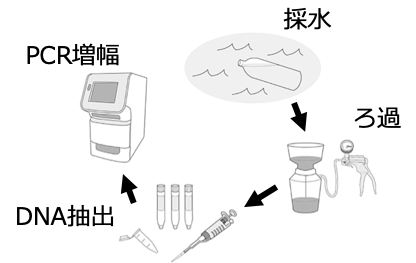
その体外に放出された水中に漂う生き物のDNAを採取することで、種を特定する技術が環境DNA分析なのです。

環境DNA手法を用いた調査は
- 水を汲むだけなので非侵襲的かつ労力の大幅な軽減
- 捕獲する必要がないので夜行性や生息密度の低い種を比較的容易に検出
- DNA 情報から生物種を同定するので形態学的な種同定の技術が必要ない
といったメリットがあります[1]。
そんな環境DNA分析には大きく2つの分析法があります。それは、
- 種特異的な検出(とある1種を対象に分析)
- 分類群全体の網羅的な検出(魚類であれば魚類全てを対象に分析)
の2つです。特に 後者を環境DNAメタバーコーディング手法と言い、私はこの環境DNAメタバーコーディング手法を用いた研究を行なっています。
どうやって水からそこにすむ生き物を特定するのか?
さて、ここからは実際にDNAから種を特定する原理(今回は②環境DNAメタバーコーディングについて)をお話ししていこうと思います。
この世のすべての生物は、自らの設計図としてDNAを持っています。DNAすなわちデオキシリボ核酸は「塩基+糖(デオキシリボース)+リン酸」(=ヌクレオチド) が鎖状に繋がってできています。
そして「塩基」には「アデニン(A)」「グアニン(G)」「シトシン(C)」「チミン(T)」の4種類があり、DNAは塩基の並び順(=塩基配列)で特徴付けられます。
したがって、塩基配列の違いがそれに応じた違いを生み出すのです。
もう少し、詳しく説明します。
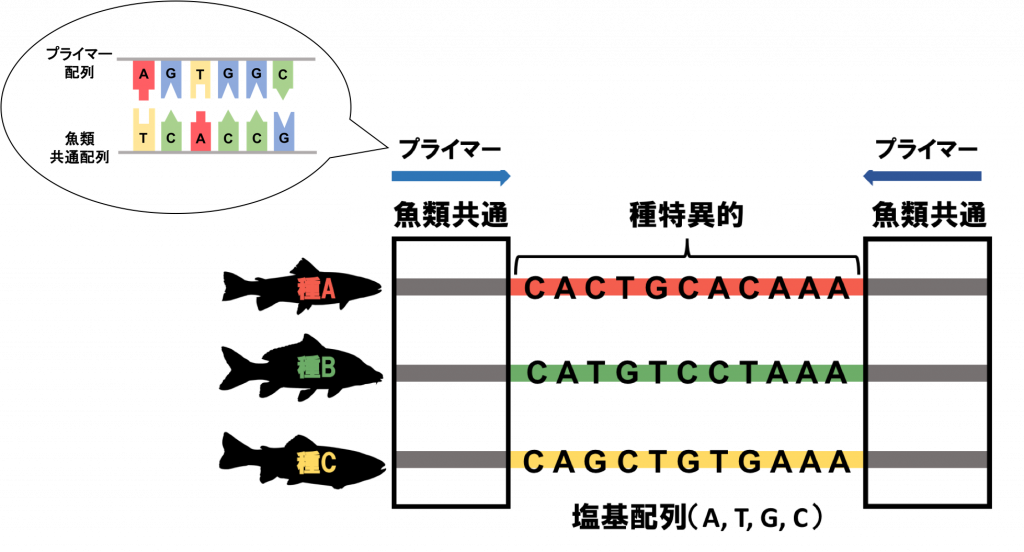
先ほども述べたように、生き物は種ごとに特異的な塩基配列を持っています。
その中で、魚類であれば皆が同じ塩基配列の並びになっている部分(下図の魚類共通部分)と、魚類の中でも種ごとに塩基配列がバラバラになっている部分(下図の種特異的部分)とがあります。
両側の魚類共通の部分に相補的な塩基配列(AにはTが、GにはCがくっつく)を人工的に作成(魚類ユニバーサルプライマー[2])し、PCRで増幅します。
PCR(ポリメラーゼ連鎖反応)についてはここでは詳しく説明しませんが、簡単に言うと、両側のプライマーに挟まれた、種ごとに異なる部分の配列をたくさん増やしてくれる操作になります。
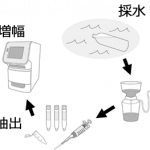
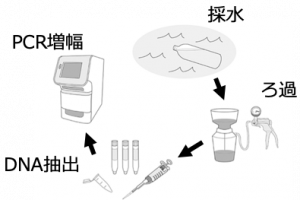
つまり、水の中に含まれる微量だった魚類の配列をものすごくいっぱいに増やしてくれる操作だと思ってください。増やした配列は、シーケンサと呼ばれる機械を用いて塩基を読み取り、塩基配列のデータベースと照合することで種を特定します。

以上のようにして、現場では水を汲むだけの作業でそこにすむ生き物を知ることができるのです。
研究背景
本当に水の中に生息する魚類は検出されるのか?
水族館で答え合わせ
千葉県立中央博物館の宮さんらは、沖縄美ら海水族館の4つの水槽水(2~10 L)から水を汲み、得られた環境DNAを分析しました。その結果、なんと4つの水槽に飼育されている魚類の9割を越す168種の検出(93.3%)に成功しています[2]。
野外の捕獲調査結果との比較
これまで、さまざまな環境DNAの研究で捕獲調査との比較が行われてきており、環境DNAメタバーコーディング手法の有用性が示されています[3, 4]。
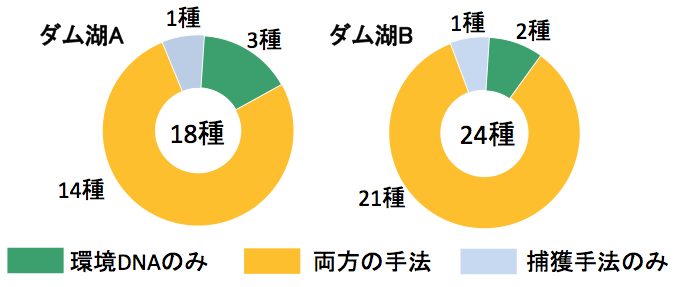
私の研究[5]においても、環境DNAメタバーコーディング手法を用いることにより、捕獲調査で得られていた9割以上の種を検出でき、かつ捕獲調査では確認されていなかった種の検出も確認されました。
どのくらい水を汲めばいいの?
では一体、水を汲むといってもどのくらい汲めばいいのでしょうか。
もちろん、より多くの地点で汲めば汲むほど、汲む回数を増やせば増やすほど検出種数は増加します。
しかし、 せっかく水を汲むだけで楽に調査できるのに、いろいろな時期にたくさんの地点で採水しなければならないのは大変です。
せっかく楽に調査できるなら楽にしたい!
そう思いますよね。
実際、 研究によって採水季節や採水位置はさまざまです。それにも関わらず、採水する季節や位置によって検出結果が異なる可能性があります。
ある対象種に着目した(種特異的)研究では、環境DNAにおける検出確率には季節性があるということも示されています[6]。したがって、魚類全体を調べる際にも、採水する季節や場所について考慮する必要があります。
未だ分かっていないこと
しかし、これまでの環境DNAメタバーコーディング手法を用いた研究では、最適なサンプリング戦略については十分に調べられていませんでした。
つまり、環境DNAメタバーコーディング手法を用いて調査する際
一体いつ, どこで, どのくらい採水すれば最小限の努力量で最大種数を検出できるのか
それはまだ明らかにされていませんでした。
私の研究内容
いつ・どこで・どのくらい水を汲めばいいのか
この疑問に答えるべく、面積や構造の異なる3つのダム湖で環境DNA調査を実施しました。
1年間を通して沖と岸の複数地点で水を汲み、そこに含まれるDNAを分析しました。全ての地点で表層水から1Lを採水しています。
最適な採水時期
まず、採水時期について検証した結果、3つのダム湖全てにおいて春に検出種数が増加しました。また、ダム湖Bについては秋にも検出種数が増加しました。
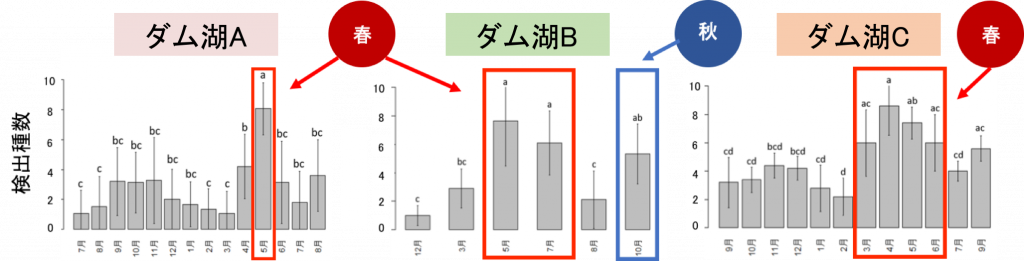
この結果は生息する多くの魚種の繁殖期に対応しており、繁殖期になると活動量が増えることで、環境DNAが放出・検出されやすくなったと考えられます。
ダム湖Bのみ、秋にも検出種数は増加していますが、これは秋に繁殖期を迎えるサケ科の魚類が多く生息しているためと考えられます。
最適な採水場所
次に、採水位置について検証した結果、ダム湖A, Bについては沖より岸で採水する方がより多くの種を検出でき、ダム湖Cについては沖と岸どちらで採水しても検出種数は変わらない結果となりました。
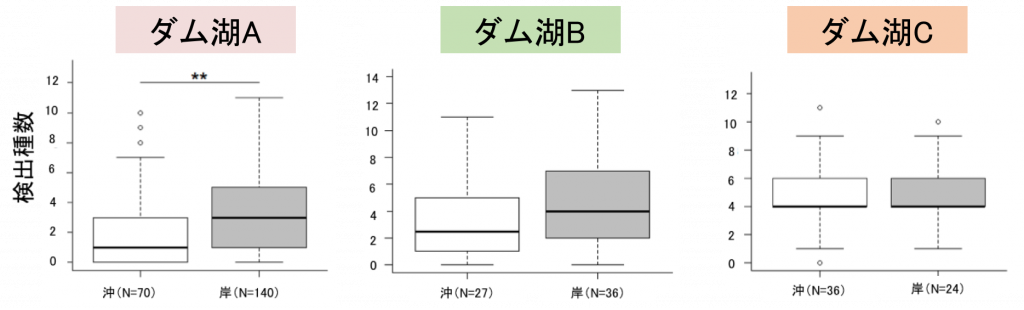
2つのダム湖において、沖よりも岸での検出種数が増加した理由としては、そもそも岸を好む魚種が多く(特に繁殖期)、沖の表層にはほとんど魚類が生息していない可能性があります。
また、ダム湖Cについては、他2つのダム湖と比べて湛水面積も小さく、単純な構造であったため沖と岸での検出種数に違いが見られなかったと考えられます。
最適な採水回数
そして、最後に採水回数について検証した結果、従来の捕獲調査で得られる種数と同程度の種数を検出するために必要なサンプル数は、3つのダム湖それぞれで異なる結果となりました。
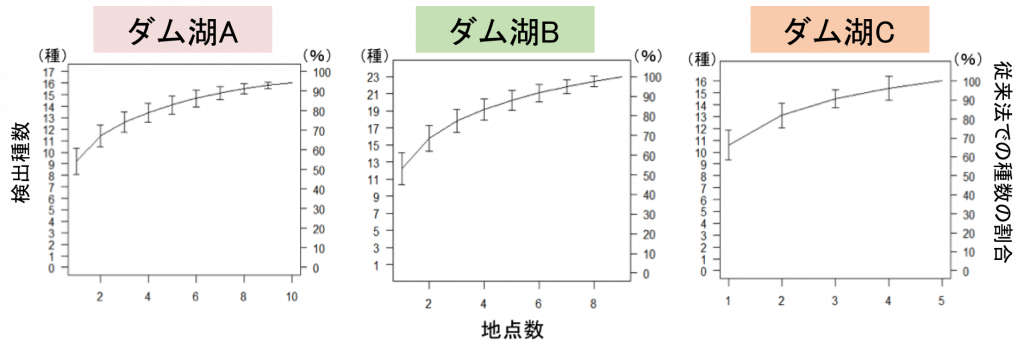
この結果は、3つのダム湖の湛水面積の違いや周囲長などの形状の違いなどと関連する可能性があります。
結論と今後の展開
以上の結果から、ダム湖において環境DNAメタバーコーディング手法を用いる際、そこに生息する魚類の繁殖期に岸で、ダム湖の形状に応じた地点数で採水することで、最小限の労力で種数を最大化できることがわかりました。
これらの結果は、今後の魚類相調査のサンプリング季節・場所・回数の決定に大きく貢献する可能性があります。
一方で、いまだに解決されていない問題もあります。
一つは、今回全てのサンプルを表層から採水しましたが、鉛直方向ではどうなっているのか、つまり「水深の違いが環境DNAの検出種数に影響を及ぼすのか」です。
これを知るためには、水深ごとに採水を行う必要があり、現在研究を進めているところです。
もう一つは、今回の調査地は止水域でしたが「流水域ではどうなるのか」ということです。
流水域では水の流れ、つまりは環境DNAの輸送についても考慮する必要があります。
こうした研究を通して、新たな水域モニタリング手法の一つとして、環境DNA手法をより社会実装化していきたいと考えています。
おわりに
以上、長くなりましたが、私の研究を紹介させていただきました。
環境DNAの魅力はわかっていただけましたでしょうか。
今回は水域、その中でも特に魚類を中心にした研究を紹介しましたが、現在の環境DNAの研究では、哺乳類や鳥類、植物など幅広い分類群に適用可能であることが示されています。
また、水からだけではなく、土壌や空気などからも生き物の環境DNAを検出することができるようになっています。
「環境DNAってすごいな」「こんな技術があるんだなあ」「こんなこともできるのではないか」など、少しでも何か感じてくださる方がいてくれたなら幸いです。
そして環境DNAには、今回紹介したもの以外にも本当にさまざな研究があります。少しでも興味を持ってくださった方は、
- 私の所属する源研究室HP: http://www2.kobe-u.ac.jp/~minamoto/index.html
- 環境DNA学会:http://ednasociety.org/
- 参考書籍:DNA情報で生態系を読み解く: 環境DNA・大規模群集調査・生態ネットワーク (生態学フィールド調査法シリーズ)
など、ぜひご覧ください。
最後までお読みいただき、ありがとうございました。
引用文献
- 高原輝彦, 山中裕樹, 源利文, 土居秀幸, & 内井喜美子. (2016). 環境 DNA 分析の手法開発の現状~ 淡水域の研究事例を中心にして~. 日本生態学会誌, 66(3), 583-599.
- Miya, M., Sato, Y. Fukunaga, T., Sado, T., Poulsen, J.Y., Sato., K., Minamoto, T., Yamamoto, S., Yamanaka, H., Araki, H., Kondoh, M., Iwasaki, W. (2015) MiFish, a set of universal PCR primers for metabarcoding environmental DNA from fishes: Detection of more than 230 subtropical marine species. Royal Society Open Science 2, 150088.
- Shaw, J. L., Clarke, L. J., Wedderburn, S. D., Barnes, T. C., Weyrich, L. S., & Cooper, A. (2016). Comparison of environmental DNA metabarcoding and conventional fish survey methods in a river system. Biological Conservation, 197, 131-138.
- Thomsen, P. F., Møller, P. R., Sigsgaard, E. E., Knudsen, S. W., Jørgensen, O. A., & Willerslev, E. (2016). Environmental DNA from seawater samples correlate with trawl catches of subarctic, deepwater fishes. PloS ONE, 11(11), e0165252.
- 速水花奈・坂田雅之・沖津二朗・片野泉・宮正樹・後藤亮・佐藤博俊・山中裕樹・源利文「ダム湖における魚類環境DNAメタバーコーディング手法の最適化」第1回環境DNA学会発表
- 今村彰生・速水花奈・坂田雅之・源利文 (2019) 河川横断構造物とニジマスが北海道のイワナ属の生息に与える影響:環境DNA分析の結果をもとに. 保全生態学研究 24, 71-81